Ein neues fluoreszentes Markerprotein haben Forscher um Professor Gerd Ulrich Nienhaus vom KIT entwickelt: Bei dem photoaktivierbaren Protein „mIrisFP“ lässt sich sowohl das Fluoreszenzlicht ein- und ausschalten als auch die Farbe des Lichts von Grün nach Rot verändern. Das Protein erlaubt dynamische Untersuchungen an Zellen und Organismen und eröffnet der zellbiologischen und molekularmedizinischen Forschung neue Möglichkeiten. In der Fachzeitschrift „Nature Methods“ stellen die Wissenschaftler nun ihre Entwicklung vor.
Fluoreszente Proteine strahlen einfallendes Licht effizient wieder ab und machen dadurch Proteine in lebenden Zellen sichtbar. Als genetisch kodierte Fluoreszenzmarker lassen sie sich für Experimente in den Lebenswissenschaften vielfältig einsetzen: Wird das Gen eines bestimmten Proteins um die Erbinformation (DNS-Sequenz) für das fluoreszierende Protein erweitert, produziert die Zelle ein so genanntes Fusionsprotein, das durch ein charakteristisches Fluoreszenzleuchten zu erkennen ist.
Zunächst war nur das grün fluoreszierende Protein (GFP) einer pazifischen Qualle bekannt. In den vergangenen Jahren entdeckte die Forschung fluoreszierende Proteine in weiteren wirbellosen Meerestieren und identifizierte unter anderem auch photoaktivierbare Proteine, deren Fluoreszenz sich durch Lichteinstrahlung gezielt steuern lässt. Die Naturformen dieser Proteine eignen sich allerdings nicht für biotechnologische Anwendungen; sie müssen dafür aufwendig optimiert werden. Bisher ließ sich bei solchen Markerproteinen entweder die Fluoreszenz ein- und ausschalten oder aber die Farbe des Lichts verändern.
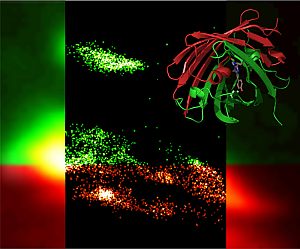
Die Forschergruppe um Gerd Ulrich Nienhaus am DFG-Centrum für Funktionelle Nanostrukturen (CFN) des KIT hat nun gemeinsam mit Wissenschaftlern der Universität Ulm, der University of Southampton/UK und der University of Illinois/USA ein als „mIrisFP“ bezeichnetes Markerprotein entwickelt. Es zeichnet sich durch die Kombination von zwei durch Licht steuerbaren Aktivierungsmodi aus: eine nicht umkehrbare Photokonversion von Grün nach Rot und eine umkehrbare Ein- und Ausschaltbarkeit der Fluoreszenz. Zudem ist mIrisFP monomer, das heißt es besteht aus nur einer Einheit, so dass es nicht zur Bildung von Proteinmolekülkomplexen kommt.
„Dadurch besitzt mIrisFP exzellente Eigenschaften für lebenswissenschaftliche Anwendungen“, erklärt Professor Nienhaus. „Besonders spannend sind sind dynamische Untersuchungen an lebenden Zellen und Organismen.“ Durch gezielte Lichteinstrahlung können bestimmte Zellen in frühen Embryonalstadien von Modellorganismen markiert und über Tage und Wochen hinweg verfolgt werden. Gezielte Markierungen von Zellkompartimenten werden mit Signalpeptiden erreicht, welche über gentechnische Modifikation auf DNS-Ebene an das fluoreszierende Protein angehängt werden. Besonders wichtig sind die erwähnten Fusionskonstrukte eines fluoreszenten Markerproteins mit einem zu untersuchenden Protein, um dieses sichtbar zu machen. Photoaktivierbare fluoreszente Proteine spielen außerdem eine Schlüsselrolle in der höchstauflösenden Fluoreszenzmikroskopie.
Mit mIrisFP lassen sich Abbildungen von Zellen mit räumlichen Auflösungen von 20 bis 30 Nanometern herstellen – weit unterhalb der Abbeschen Auflösungsgrenze von rund 200 Nanometern, die lange als physikalische Grenze galt und genauere Einblicke in die molekularen Prozesse in der lebenden Zelle verhinderte. In der Fachzeitschrift „Nature Methods“ stellt die Forschergruppe das neue Markerprotein vor und untersucht mit höchstauflösender Photoaktivierungs-Lokalisationsmikroskopie (PALM) molekulare Prozesse bei der Bewegung einer menschlichen Krebszelle. So lässt sich verfolgen, wie bestimmte mit mIrisFP sichtbar gemachte Proteine in einem Bereich der Zelle entfernt werden, um anschließend in einem anderen Bereich der Zelle wiederum in neue Strukturen eingebaut zu werden.
Literatur
A photoactivatable marker protein for pulse-chase imaging with superresolution. Jochen Fuchs, Susan Boehme, Franz Oswald, Per Niklas Hedde, Maike Krause, Jörg Wiedenmann & G Ulrich Nienhaus. Nature Methods. Published online: 4 July 2010; | doi: 10.1038/nmeth.1477.
http://dx.doi.org/_10.1038/nmeth.1477
Im Dialog mit der Gesellschaft entwickelt das KIT Lösungen für große Herausforderungen – von Klimawandel, Energiewende und nachhaltigem Umgang mit natürlichen Ressourcen bis hin zu Künstlicher Intelligenz, technologischer Souveränität und demografischem Wandel. Als Die Universität in der Helmholtz-Gemeinschaft vereint das KIT wissenschaftliche Exzellenz vom Erkenntnisgewinn bis zur Anwendungsorientierung unter einem Dach – und ist damit in einer einzigartigen Position, diese Transformation voranzutreiben. Damit bietet das KIT als Exzellenzuniversität seinen mehr als 10 000 Mitarbeitenden sowie seinen 22 800 Studierenden herausragende Möglichkeiten, eine nachhaltige und resiliente Zukunft zu gestalten. KIT – Science for Impact.